ARTÍCULO ORIGINAL
Variabilidad morfológica y molecular en genotipos de algodón (Gossypium hirsutum L.) de tres orígenes geográficos diferentes
Morphological and molecular variability in cotton (Gossypium hirsutum L.) genotypes from three geographical origins
Variabilidade morfológica e molecular em genótipos de algodão (Gossypium hirsutum L.) de três origens geográficas diferentes
Klein, Lorena Marina1; Etchart, Valeria Juliana 3; Tcach,
Mauricio Alfredo1; Gonzales, Ariela Judith 1; Tcach,
Nidya Elisa1; Dileo, Pablo Nahuel2; Spoljaric, Monica
Viviana1
1 EEA INTA Sáenz Peña, Ruta
Nacional Nº 95 Km 1108, 3700 Pcia. R. Sáenz
Peña, Chaco, Argentina
2 EEA.INTA Reconquista, Ruta Nacional Nº 11 Km
773, 3567. Reconquista, Santa Fe, Argentina
3 Instituto de Genética “Ewald A. Favret”
CICVyA-INTA, CC25 (1712) Castelar, Buenos Aires, Argentina
DOI: https://doi.org/10.35305/agro40.e025
Resumen
El algodón (Gossypium hirsutum L.) ha perdido variabilidad debido a su proceso de domesticación. Por esta razón, es necesaria la caracterización molecular y fenotípica de nuevos materiales para los programas de mejoramiento genético de esta especie. El objetivo de este trabajo fue caracterizar la variabilidad de 18 accesiones de tres orígenes geográficos Argentina, Estados Unidos y China por características genéticas y fenotípicas. Los materiales pertenecen al Banco de germoplasma de INTA y se utilizan en el programa de mejoramiento. Para el estudio genético se utilizaron 16 pares de oligonucleòtidos microsatélites. En la caracterización fenotípica se utilizaron variables asociadas al mapeo en dos campañas agrícolas (2015/2016 y 2016/2017) realizadas en el campo de INTA EEA Sáenz Peña. Los resultados genéticos mostraron valores medios/altos de polimorfismo (62,22%) y en el dendrograma se observaron dos grupos; uno representado por Stoneville 474 (Estados Unidos) y ocho genotipos de China y un segundo grupo formado por Deltapine 16 (Estados Unidos), los genotipos de Argentina y un genotipo de China. Los resultados fenotípicos en un análisis multivariado de Componentes Principales entre campañas agrícolas presentaron diferencias significativas, mientras que fueron no significativas entre los diferentes orígenes geográficos. Se concluye que existe uniformidad en los caracteres morfológicos en los tres orígenes geográficos y diferenciación genética a partir del análisis molecular siendo las accesiones BGSP 756, BGSP 767 y BGSP 783 de China las que presentaron mayores distancias con respecto a los materiales de Argentina y Estados Unidos; justificando la introducción de germoplasma de origen de China al programa de mejoramiento de INTA.
Palabras claves: algodón; variabilidad genética; microsatélites; mapeo de plantas
Abstract
Cotton (Gossypium hirsutum L.) has lost variability due to its domestication. For this reason, it is necessary to obtain the molecular and phenotypic characterization of new breeding materials for this species. The objective of this work was to characterize the genetic and phenotypic variability of 18 accessions from Argentina, the United States, and China. The materials belong to the INTA (Argentina’s Agricultural Technology Institute) genebank and are used for breeding programs. Sixteen pairs of microsatellite oligonucleotides were used for the study. For the phenotypic characterization, variables associated with mapping were used in two cropping seasons (2015-2016 and 2016-2017) in the INTA experimental field in Sáenz Peña, province of Chaco, Argentina. The genetic results showed medium/high values of polymorphism (62.22%), and the dendrogram showed two groups: one made up of Stoneville 474 (United States) and eight genotypes from China, and a second group made up of Deltapine 16 (United States), the genotypes from Argentina, and one genotype from China. The phenotypic results obtained through Principal Components Analysis showed significant differences between cropping seasons, while no significant differences were detected between geographical origins. The morphological characters of accessions from the three different geographical origins were similar. However, genetic differentiation was found between the microsatellites analyzed, with accessions BGSP 756, BGSP 767 and BGSP 783 from China exhibiting higher genetic distances than materials from both Argentina and the United States. Our results support the introduction of cotton germplasm from China for INTA's breeding program.
Keywords: cotton; genetic variability; microsatellites; plant mapping
Resumo
O algodão (Gossypium hirsutum L.) perdeu variabilidade devido ao seu processo de domesticação. Por esta razão, a caracterização molecular e fenotípica de novos materiais é necessária para os programas de melhoramento genético desta espécie. O objetivo deste trabalho foi caracterizar a variabilidade de 18 acessos de três origens geográficas Argentina, Estados Unidos e China por características genéticas e fenotípicas. Os materiais pertencem ao Banco de Germoplasma do INTA e são utilizados no programa de melhoramento. Para o estudo genético, foram utilizados 16 pares de oligonucleotídeos microssatélites. Na caracterização fenotípica, foram utilizadas variáveis associadas ao mapeamento em duas campanhas agrícolas (2015/2016 e 2016/2017) realizadas no campo do INTA EEA Sáenz Peña. Os resultados genéticos mostraram valores de polimorfismo médio/alto (62,22%) e no dendrograma foram observados dois grupos; um representado por Stoneville 474 (Estados Unidos) e oito genótipos da China e um segundo grupo formado por Deltapine 16 (Estados Unidos), os genótipos da Argentina e um genótipo da China. Os resultados fenotípicos numa análise multivariada dos Componentes Principais entre campanhas agrícolas, apresentaram diferenças significativas, enquanto entre as diferentes origens geográficas não foram significativas. Conclui-se que existe uniformidade nos caracteres morfológicos nas três origens geográficas e diferenciação genética a partir da análise molecular, sendo os acessos BGSP 756, BGSP 767 e BGSP 783 da China os que apresentavam maiores distâncias em relação aos materiais da Argentina e dos Estados Unidos; justificando a introdução de germoplasma de origem chinesa no programa de melhoramento do INTA.
Palavras-chave: algodão; variabilidade genética; microssatélites; mapeamento de plantas
Introducción
La planta de algodón produce la fibra
textil natural más importante del mundo y pertenece al género Gossypium,
que incluye aproximadamente 50 especies, de las cuales 45 son diploides (2n =
2x = 26) y 5 tetraploides (2n = 4x = 52) (Wendel et al., 2010).
Cuatro de ellas se cultivan en más
de 65 países principalmente para aprovechar su fibra: G. herbaceum, G.
arboreum (ambas diploides portadoras de genoma A), G. hirsutum,
Upland o americano y G. barbadense Pima o egipcio (ambas tetraploides
portadoras de genoma AD). Se ha postulado que el algodón alotetraploide
evolucionó aproximadamente hace 1.5 millones años, a partir de un único evento
de hibridación entre G. herbaceum (genoma A1) y G. raimondii (genoma D5),
seguido de una diploidización, siendo esto quizás uno de los principales
motivos del bajo nivel de polimorfismo encontrado en el algodón cultivado (Endrizzi et al., 1985, Wendel et al., 1992;
Brubaker et al., 1999). Asimismo, la domesticación de G.
hirsutum surgió hace 5000 años en la península de Yucatán (Wendel
et al., 1992). La historia del algodón Upland, que actualmente se cultiva
en el 90% de la superficie algodonera en el mundo, comienza con las introducciones
de algodones de las tierras altas de México por los Estados Unidos a principios
de 1800 (Jenkins, 2003; Rai et al.,
2013; Wendel et al., 1992). Posteriormente diversas
introducciones fuera de los EE.UU. se incorporaron a diferentes programas de
reproducción para el desarrollo de variedades adaptadas. En este sentido el
algodón Upland derivó de cuatro categorías generales de variedades con un
ancestro común: Acala, Stonville, Coker y Deltapine (Niles,
1980). Algunos autores sostienen que la diversidad genética de Upland es
muy baja debido a una selección artificial intensiva en características de
importancia económica durante la domesticación y procesos de mejora (Abdalla
et al., 2001; Wendel et al., 1992). Estudios con
marcadores moleculares revelan esta tendencia a
partir de que se ha encontrado un mayor número de fragmentos polimórficos
amplificados en algodón tipo silvestre en relación a genotipos sometidos a
procesos de mejora (Lori et al., 2016). Por otro lado, es
importante tener en cuenta que los diferentes programas de mejoramiento genético
de algodón generan presiones de selección en adaptación a ambientes locales y
adversos, proceso por el cual se han formado grupos de germoplasma distintivos
(Lori et al., 2016). En este sentido, Tyagi et al. (2014) examinaron mediante marcadores microsatélites (SSR:
Secuencia Simples Repetidas) variedades de algodón Upland que abarcaban un
siglo de cría en los Estados Unidos y encontraron que los germoplasma se
agruparon por regiones formando 4 grupos: 1)
Oriental: Carolina del Norte y del Sur, Georgia y Alabama, asociado al
desarrollo de germoplasma con buena calidad de fibra y resistencia a
enfermedades como el marchitamiento (PeeDee, Coker, Meade, Auburn, etc.); 2)
Centro Sur: tipos Delta, programas de mejoramiento para el Delta del
Mississippi y las áreas circundantes de Arkansas y Luisiana (Deltapine,
Stoneville, Delfos, DES, Dixie King, etc.); 3) Llanuras: programas para los
ambientes de llanuras de Texas y Oklahoma que incluyeron características
prominentes como cápsulas resistentes a tormentas, precocidad y un grado de
tolerancia a la sequía (Tamcot, Rowden, Lockett, Lankart, Paymaster, etc.) y
4); Occidental: programas para algodones de alta producción de fibra bajo riego
controlado, se centraron en California, Nuevo México y Arizona (Acala,
Germains, Hopi, etc.). Fang et al. (2013), también
utilizaron marcadores SSR para examinar las relaciones entre cultivares de
secano provenientes de distintos países. Estos resultados indicaron la
formación de 15 grupos en base a la historia de cría y las genealogías.
Asimismo, Chen y Du Xiong (2006) estudiaron la diversidad
genética del germoplasma de algodón en China, siendo las introducciones del
extranjero su principal fuente de germoplasma base (DPL, Stonville, King,
Uganda, Foster y Trice). Esta diversidad fue analizada por SSR en base a tres
períodos de cría (antes de 1950; 1950-1960; 1960-1970), tres áreas de cultivo
(Valle del río Yangtze; Valle del río Huanghe, y norte de China) y en dos
grupos de germoplasma, los desarrollados en China y los extranjeros (América,
Rusia, Uganda). Este estudio les permitió determinar que la diversidad a nivel
del genoma del germoplasma base del segundo y tercer período de reproducción
disminuyó en comparación con el primer período, lo que indica que el fondo
genético del algodón de China se ha estrechado gradualmente. En Argentina, un
estudio similar utilizando marcadores moleculares SSR fue reportado por Gómez (2007), donde se estimó la
diversidad genética presente en una colección núcleo conservada por el Banco
Activo de Germoplasma de INTA en base al origen geográfico, datos agronómicos y
a su importancia histórica en el desarrollo de variedades. Los resultados
indicaron que las variedades INTA (Argentina) fueron más similares a las
variedades Reba (Paraguay), Deltapine, Stoneville (EE.UU.) y australianas que a
las variedades Acala (EE.UU.) reflejando así la historia del mejoramiento del
cultivo en el país. Además, un número reducido de entradas de G. hirsutum
L. presentaron alelos específicos lo que limita la ampliación de la diversidad
alélica y el potencial de mejoramiento del cultivo de algodón (Gómez,
2007).
La historia del algodón en Argentina comenzó en el siglo XVIII, en las misiones jesuíticas a lo largo del río Bermejo y en Misiones (Girbal-Blacha, 2012). En la década de 1920 se sembraron las primeras hectáreas en el Chaco, con algodones denominados “tipo Chaco”, derivados de variedades introducidas de Luisiana (EE.UU.) de gran irregularidad en los rendimientos debido a que se obtenían a partir de sucesivos cruzamientos naturales a campo y mezclas de semillas de desmotadoras. Años después comenzaron a sembrarse genotipos selectos sobre germoplasma introducido desde los Estados Unidos, como Deltapine, Stoneville, Acala y Coker Wilds. En el año 1956 el programa de mejoramiento del INTA emitió la primera variedad nacional llamada SP Toba INTA, con base genética de Deltapine y Stoneville (Royo et al., 2006). A partir de la combinación interespecífica H.A.R (G. hirsutum L.; G. arboreum L.; G. raimondii Ulbrich) desarrollada por el Institute de Reserche du Coton et des Textiles Exotiques (1946), en 1969 se obtiene la variedad Chaco 510 INTA, con rasgos de precocidad y calidad de fibra superior. Cinco años más tarde se combinan las características de Chaco 510 INTA y la variedad de origen de Paraguay, denominada Reba P279, de elevado potencial de producción, dando lugar a la obtención y difusión de variedades como Guazuncho INTA (1982), Quebracho INTA (1982) y Pora INTA (1982), como así también Guazuncho 2 INTA (1989), Gringo INTA (1989), Chaco 520 INTA (1994), Cacique INTA (1994) y Oro Blanco INTA (1999). Recientemente se destacan Guazuncho 3 INTA (2004), Chaco 530 INTA (2004), Oro Blanco 2 INTA (2004), Poraite INTA (2008) y Gualok INTA (2008).
Por ultimo en año 2019, INTA introdujo al mercado tres nuevas variedades de algodón: Guarani INTA BGRR, Pora 3 INTA BGRR, Guazuncho 4 INTA BGRR, las cuales cuentan con la tecnología BG/RR (desarrollado por Monsanto) que le confiere a la planta protección a ciertos insectos lepidópteros y tolerancia a las aplicaciones de herbicida presentando además elevado potencial productivo y plasticidad a condiciones ambientales (Gensus Genetica Sustentable).
La introgresión de eventos biotecnológicos y de mutagénesis, así como el intercambio de germoplasma de diferentes orígenes geográficos, han resultado una herramienta de importancia para incrementar la variabilidad genética. Por lo cual, la incorporación en el año 2010 de nuevas entradas de algodón de origen de China al Banco de Germoplasma del INTA posibilita la utilización de nuevas fuentes de variabilidad genética no consideradas hasta el momento. En la actualidad, la diversidad alélica es de gran importancia en los programas de mejoramiento ya que éstos necesitan introducir nuevos alelos al pool de líneas elite para obtener un mayor progreso genético. En este sentido, se plantea la necesidad de estudiar la variabilidad existente entre los germoplasmas de algodón provenientes de tres regiones geográficas diferentes a partir de una caracterización genética y fenotípica.
Materiales y métodos
Material vegetal:
Se evaluaron 18 accesiones de algodón (Gossypium hirsutum L.) de tres regiones geográficas pertenecientes a la colección de algodón del Banco de Germoplasma y Mejoramiento Genético de INTA Sáenz Peña, Chaco (Argentina). Todos fueron caracterizados genotípica y fenotípicamente. En la evaluación genotípica se incorporó a Gossypium barbadense (silvestre) como outgroup. El listado de materiales con su designación, origen y características se describen en la Tabla 1.
Tabla 1. Genotipos de algodón pertenecientes al
Banco de Germoplasma de EEA Sáenz Peña.

1 - Caracterización genotípica
Extracción de ADN:
Los estudios se llevaron a cabo en el Laboratorio de Biotecnología de la EEA INTA- Sáenz Peña. Se analizó un total de 95 muestras (18 accesiones y un silvestre con 5 plantas cada uno, propiciando de repeticiones). Se extrajo ADN genómico a partir de hojas jóvenes frescas, empleando el kit “illustra Nucleon PhytoPure Genomic DNA (GE Healthcare Life Sciences U.K.) y siguiendo el protocolo descripto por el fabricante.
Cuantificación y evaluación de la calidad del ADN extraído:
La calidad, integridad y concentración de ADN se determinó mediante electroforesis en geles de agarosa al 0,8 % (p/v), preparados con buffer Tris-Borato-EDTA (TBE) 0,5X y posterior tinción con GelGreen® en una concentración final de 3X. Se utilizó ADN del fago Lambda (λ) cortado con la enzima de restricción HindIII como marcador de masa (Thermo Scientific).
Amplificación de los microsatélites (SSR), separación y visualización de los productos de amplificación:
La caracterización molecular se realizó empleando un total de 39 marcadores SSR (Tabla 2) de secuencia pública, obtenidos de la base de datos CottonGen (Yu, J. et al., 2021)
Las reacciones de amplificación de todos los primers y tiempos de ciclado, se llevaron a cabo según los protocolos descriptos por Etchart (2009).
Los productos de la amplificación se separaron en geles desnaturalizante de poliacrilamida 6% (Acrilamida: bisacrilamida 19:1; 7 M urea) de 0,4 mm de espesor. En cada gel se incluyó un marcador de peso molecular de 100pb (GeneRuler 100 pb-Thermo Scientific) y la visualización de las bandas se realizó por tinción con nitrato de plata y revelado con carbonato de sodio.
Tabla 2. Marcadores SSR obtenidos a partir de la base de datos
CottonGen.

Análisis estadístico:
Los datos fueron analizados mediante el programa estadístico Info-Gen versión 2016 (Balzarini, M. y Di Rienzo, J., 2016). Según lo descripto por Botstein et al. (1980), un marcador es altamente informativo cuando presenta valores de PIC mayores a 0,5, moderadamente informativo para valores entre 0,5 y 0,25 y levemente informativo para valores menores a 0,25. Otros parámetros analizados fueron: el número de bandas polimórficas (BP) y monomórficas (BM), como así también número de bandas totales (BT), y la probabilidad de que 2 individuos compartan el mismo alelo por azar (PDICMA).
Análisis de conglomerados: los datos moleculares obtenidos fueron transformados mediante el índice de similitud de Jaccard y la fórmula de transformación
![]()
para obtener la matriz de distancia cuyos valores se establecen entre 0 y
1 (menor distancia y mayor distancia respectivamente).
Para representar la relación existente entre las muestras evaluadas se realizó el análisis de agrupamiento mediante el método de encadenamiento promedio (Unweighted Pair Group Method with Arithmetic Mean: UPGMA).
2 - Caracterización fenotípica
Los ensayos a campo fueron conducidos en INTA (Instituto Nacional de Tecnología Agropecuaria), EEA (Estación Experimental Agropecuaria) Sáenz Peña, (Latitud Sur 26º 47' 27" y Longitud Oeste 60º 26' 29"; Altitud 90 m.s.n), Chaco (Argentina).
Diseño experimental:
Se utilizó un diseño en bloques completos aleatorizado con 3 repeticiones. El primer ensayo se sembró el 23 de noviembre de 2015 y el segundo el 21 de noviembre de 2016 con espaciamiento entre hileras de 1 m, una densidad de 5 plantas por metro. El control de malezas y plagas fueron realizados según las prácticas recomendadas por PROCALGODON (2008).
Condiciones climáticas:
El área de estudio se caracteriza por ser un clima subtropical con estación seca. El semestre más caluroso va de octubre (mín.16ºC) a marzo (máx. 34,2ºC en enero) con precipitaciones entre 1.000 a 600 mm anuales decreciendo de Este a Oeste siendo la evapotranspiración potencial anual de 1600 mm.
En la campaña 2015/16 los valores de temperatura máxima promedio, temperatura mínima promedio y de lluvias promedio fueron de: 32,9°C, 20,7°C y 384,3 mm, respectivamente. Mientras que para la campaña 2016/17 la temperatura máxima promedio fue de 32,1°C, la temperatura mínima promedio fue de 20,5°C, y el promedio de lluvias fue de 543,4 mm. Valores registrados a partir de la fecha de siembra hasta cosecha (31 de marzo de 2016 y 2017).
Variables
Se midieron las variables asociadas al mapeo a fin de ciclo (130 días), seleccionando en un metro lineal 5 plantas por bloque y genotipo. Las mismas fueron: Altura de Planta (AP, cm), Ramas Vegetativas (RV), Nº de Nudos del tallo principal (NN), Longitud media de los Entrenudos del tallo principal (LME), Nº de Nudo del tallo principal en que se inserta la Primer Rama Fructífera (NPRF), Nº de Frutos sobre ramas Vegetativas (FV), Nº de Ramas Fructíferas (NRF), Total de Puntos Fructíferos de la planta (TPF), Porcentaje de Frutos fijados del total de puntos diferenciados en la planta (Fij%), Nº frutos Caídos (CAI), Nº de Capullos (CAP), nudo vegetativo (NV) y N° de frutos o bochas (Boch).
Análisis estadístico:
Se realizó un análisis multivariado con el programa R versión 3.4.3 (R, 2011) el método HCPC (Hierarchical Clustering on Principal Components) utilizando el paquete FactoMineR. En primer lugar, se estandarizaron los datos y se construyó una matriz de correlaciones lineales para un ACP (Análisis de Componentes Principales) con los datos colectados de ambas campañas (2015/2016 y 2016/2017). En segundo lugar, se procedió al análisis de la correlación de la variable categórica (Campañas) con las Componentes Principales (CP) utilizando las elipses de confianza a un nivel de significación del 5%. Una vez verificado las diferencias de medias entre grupos (campañas) y origen geográfico, se procedió a realizar nuevamente ACP para cada campaña.
Resultados
1 - Caracterización genotípica
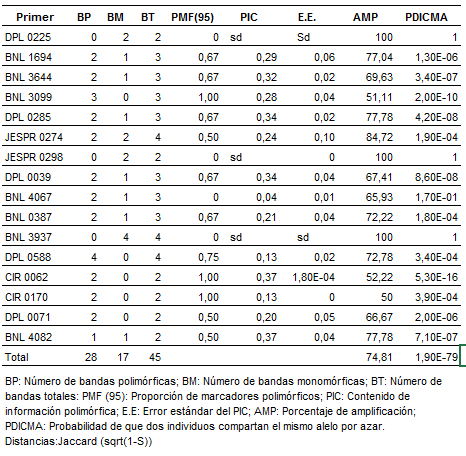
Del total de los 39 marcadores microsatélites, 16 produjeron patrones de amplificación claros y reproducibles que permitieron caracterizar las accesiones con precisión. Trece resultaron polimórficos y 3 monomórficos entre las muestras analizadas. El número de fragmentos amplificados por marcador varió entre 2 y 4, generando un total de 45 fragmentos analizados, donde 17 (38%) fueron monomórficos y 28 (62 %) polimórficos. Los valores de PIC oscilaron entre 0,37 y 0,04. Los mayores valores de contenido de información polimórfica (PIC) se evidenciaron para los marcadores BNL 4082, CIR 0062, DPL 0285, DPL 0039, BNL3644, BNL1694 y BNL 3099 cuyos valores se encuentran entre 0,37 y 0,28, resultando ser marcadores moderadamente informativos según el rango de 0 a 0,5 establecido por Balzarini et al. (2010), los cuales podrían ser de utilidad en posteriores estudios de caracterización (Tabla 3). La menor probabilidad de que 2 individuos compartan el mismo alelo se evidenció para el marcador CIR 0062, mientras que DPL 0225, JES 0298 y BNL 3937 presentan los menores valores de capacidad discriminatoria.
Tabla 3:
Estadística descriptiva del polimorfismo observado mediante 16 marcadores SSR
utilizados para caracterizar genotípicamente 18 accesiones provenientes de tres
regiones geográficas.
Análisis de conglomerados:
El análisis de agrupamiento mediante UPGMA mostró la formación de tres grupos:
Silvestre
(outgroup)
Stoneville 474 y genotipos de China
Deltapine 16 y genotipos de Argentina.
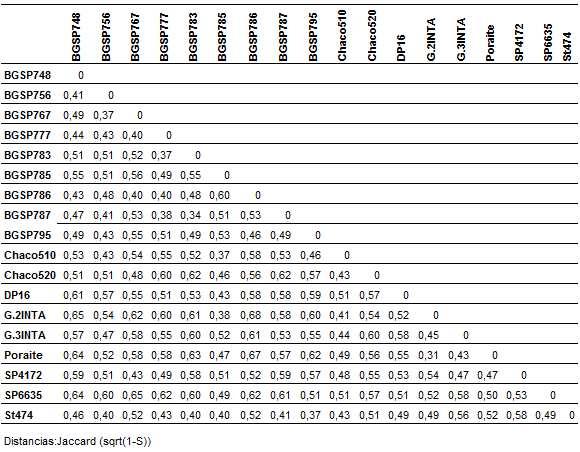
El primer grupo estuvo conformado por Stoneville 474 (EE.UU.) y ocho de los nueve genotipos provenientes de China caracterizados (BGSP 787, BGSP 777, BGSP 783, BGSP 795, BGSP 756, BGSP 748, BGSP 786, BGSP 767), siendo los germoplasmas BGSP 787 y BGSP 783 los que presentaron menor distancia. El segundo grupo formado por Deltapine 16 (EE.UU.) y los genotipos provenientes de Argentina (a excepción de BGSP 785 de China), mostrando menor distancia Poraite INTA y Guazuncho 2 INTA. Por otra parte, el germoplasma de China BGSP 785 se encuentra muy asociado al germoplasma argentino Chaco 510 INTA (Figura 1). En la Tabla 4 se muestra que BGSP 786 presenta mayor distancia en relación al genotipo Guazuncho 2 INTA (68%), Poraite INTA (67%), SP 6635 (62%) y Guazuncho 3 INTA (61%). Para BGSP 767 la mayor distancia es con SP 6635 (65%) y Guazuncho 2 INTA (62%). Para BGSP 748 la mayor distancia se observa con Guazuncho 2 INTA (65%), Poraite y SP 6635 (64%). Para BGSP 783 la mayor distancia se observa Poraite (63%), Chaco 520 INTA (62%), Guazuncho 2 INTA (61%), SP 6635 y Guazuncho 3 INTA (60 %). Para BGSP 777 la mayor distancia se observa con SP 6635 (62%), Guazuncho 2 INTA y Chaco 520 INTA (60%). Para BGSP 787 la mayor distancia se encuentra con Chaco 520 INTA (62%) y SP 6635 (61%). Para BGSP 795 la mayor distancia es con Poraite INTA (62%) y Guazuncho 2 INTA (60%). Por último, para BGSP 756 la mayor distancia es con SP 6635 (60%).

Figura 1: Dendrograma
para las 18 accesiones
provenientes de tres regiones geográficas y un genotipo silvestre utilizando distancia de
Jaccard, y método de encadenamiento UPGMA.
Tabla 4: Matriz de distancia obtenido a partir de las 18 accesiones
provenientes de tres regiones geográficas, basada en el índice de similitud de
Jaccard.
2 - Caracterización fenotípica
Análisis de Componentes Principales en las dos campañas agrícolas:
Las variables con mayor coordenada en la CP1 (Componente Principal 1) fueron la AP, NN, NRF, LME, CAI y TPF. Para la CP2 (Componente Principal 2), las variables de FV, Boch y Fij%(Tabla 5). La correlación significativa (Elipses de confianza=Alfa=0,05) de la variable categórica campañas agrícolas, en relación a las CP1 y CP2 presentaron diferencias significativas (Figura 2). La primera campaña se sitúa en el plano de la CP1 hacia la izquierda, es decir que toma valores negativos para las variables. Contrariamente fue el comportamiento de la segunda campaña, situándose en el plano de la CP1 hacia la derecha, es decir valores positivos. Los caracteres fenotípicos de mayor importancia para diferenciar a las campañas agrícolas fueron las variables relacionadas al crecimiento en AP, NN, NRF, LME y reproductivas CAI, TPF. La Figura 3 muestra la falta de correlación significativa (Elipses de confianza= Alfa=0,05) de la variable categórica origen geográfico con las componentes en ambas campañas ya que cuando las elipses de confianzas se interceptan no hay significado entre los orígenes geográficos.
Tabla
5: Coordenadas
de las variables de mapeo con las CP1 y CP2 para primera y segunda campaña.


Figura 2. Correlación
significativa (Elipses de confianza= Alfa=0,05) de la variable categórica
campaña agrícola con las componentes.

Figura 3. Correlación significativa
(Elipses de confianza= Alfa=0,05) de la variable categórica origen geográfico
con las componentes.
Agrupamiento jerárquico en componentes principales por campaña agrícola:
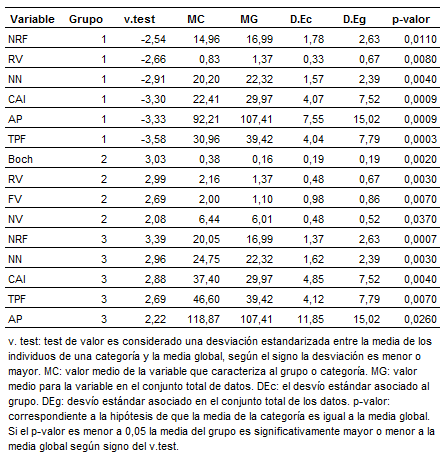
Una vez verificado las diferencias en el comportamiento entre las campañas y las no diferencias entre los orígenes geográficos se realizó un ACP (Análisis de Componentes Principales) y posteriormente un análisis de los grupos por campaña. En Tabla 6 se presentan los grupos de la primera campaña caracterizados en función de las variables de mapeo. Los genotipos del grupo 1 (Guazuncho 2 INTA, BGSP 786, BGSP 787, SP 6635, Guazuncho 3 INTA, Stonville 474 y Poraite INTA) se caracterizaron por presentar promedios más bajo en las variables de NRF, RV, NN, AP, CAI y TPF en relación a la media general de todos los genotipos. Un comportamiento diferente caracterizó al grupo 2 (BGSP 795, SP 4172, BGSP 748, BGSP 767, BGSP 783 y BGSP 756). El mismo presentó los promedios más altos en las variables de Boch, FV, RV y NV en relación a la media general. Por último, el grupo 3 (Chaco 520 INTA, BGSP 785, Chaco 510 INTA, Deltapine 16 y BGSP 777) presentó los valores más altos en promedio de NRF, RV, NN, AP, CAI y TPF en relación a la media general.
Tabla 6. Descripción de los grupos a través de
las variables morfológicas para la primera campaña.
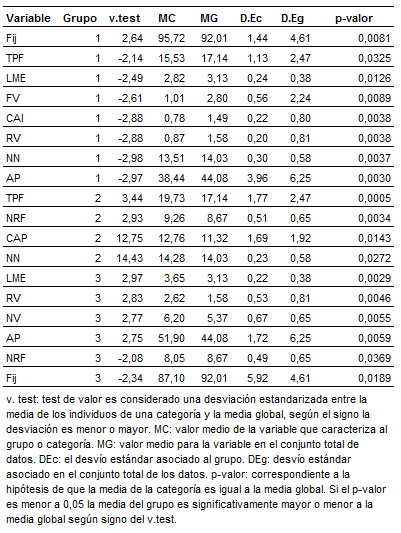
En Tabla 7 se presentan los grupos de los genotipos caracterizados en función de las variables de mapeo para la segunda campaña. Los genotipos del grupo 1 (Chaco 510 INTA, Deltapine 16, BGSP 795, Guazuncho 2 INTA, Poraite INTA, Stonville 474, Guazuncho 3 INTA) se caracterizaron por presentar promedios más bajo en las variables de Fij%, TPF, LME, FV, CAI, RV, NN y AP en relación a la media general de todos los genotipos. El grupo 2 (BGSP 787, SP4172, SP 6635, BGSP 777, BGSP 748, BGSP 786, Chaco 520 INTA) presentó los promedios más altos en las variables de TPF, NRF, CAP y NN en relación a la media general. Por último, el grupo 3 (BGSP 785, BGSP 756, BGSP 767, BGSP 783) presentó los valores más alto en promedio de LME, RV, NVy AP en relación a la media general y valores más bajos que la media general de NRF y Fij%.
Tabla 7. Descripción de los grupos a través de las
variables morfológicas para la segunda campaña.
La diversidad presente en el algodón cultivado Upland es muy baja, debido a una selección artificial intensiva para caracteres económicos durante la domesticación y proceso de cría (Abdalla et al., 2001; Wendel et al., 1992). Fenómeno similar se ha observado en otros cultivos como maíz (Zea mays L.) (Vigouroux et al., 2005), arroz (Oryza sativa L.) (Garris et al., 2005), girasol (Helianthus annuus L.) (Liu y Burke., 2006) y soja (Glycine spp.) (Lam et al., 2010; Li et al., 2010). Una estrategia para aumentar la diversidad en los programas de mejoramiento genético es mediante el uso de genotipos silvestres. Sin embargo, hay restricciones relacionadas con barreras para el cruzamiento (Pereira et al., 2012) como así también las desventajas que conlleva la incorporación de rasgos muy diferentes a los requeridos para la producción comercial de algodón. Por esta razón, la mejora de la producción de algodón se realizó generalmente desde una base genética estrecha. En este contexto, la introducción de nuevo germoplasma puede resultar de utilidad para aumentar la variabilidad en una población de mejora. En Argentina, los cultivares generados por el programa de mejoramiento genético de algodón de INTA EEA Sáenz Peña, presentan similitud o menor distancia genética con las Stonville y Deltapine y se diferencian principalmente de las variedades tipo Acala (Gómez, 2007). Sin embargo, no hay antecedentes en la utilización de germoplasma de origen geográfico de China en la generación de nuevos cultivares. Por lo tanto, el conocimiento de la variabilidad genética de nuevos genotipos ingresados en el año 2010 al Banco de Germoplasma de INTA es clave para la decisión de incorporarlos al proceso de mejora.
La variabilidad genética estudiada entre los 18 genotipos de diferentes orígenes geográficos (China, Estados Unidos y Argentina) evaluados en este trabajo, mostraron valores medios-altos de polimorfismo (62,22%), y un número de fragmentos amplificados de 1 a 4. Estos resultados son similares con los reportados por Gómez (2007) en la estimación de la diversidad genética mediante marcadores microsatélites en entradas de algodón (Gossypium hirsutum L.) del Banco de Germoplasma de INTA.
A nivel molecular los genotipos de los tres orígenes geográficos se dividieron en dos grandes grupos (Figura 1). Los genotipos de origen geográfico de Argentina presentaron menor distancia genética con Deltapine 16 (EE.UU.), coincidiendo con lo hallado por Gómez (2007) cuando analizo una colección núcleo del Banco de germoplasma de INTA, concluyendo que la base genética de las variedades INTA esta influenciadas principalmente por las introducciones de germoplasma provenientes de los Estados Unidos de América (Deltapine, Stoneville, Acala y Coker Wilds), (Gómez, 2007); mientras el origen geográfico de China presento menor distancia genética con Stonville 474 (EE.UU.). La principal fuente de germoplasma base de China fueron las introducciones del extranjero especialmente Deltapine, Stonville, King, Uganda, Foster y Trice (Chen y Du Xiong, 2006). Además, es importante tener en cuenta que los diferentes programas de mejoramiento genético de algodón generan presiones de selección en adaptación a ambientes locales y adversos, proceso por el cual se genera la formación de grupos de germoplasma distintivos (Lori et al., 2016). Djaboutou et al. (2017) reportaron observaciones similares en estudios de variabilidad y heredabilidad de caracteres morfológicos en 14 genotipos de diferentes países pertenecientes al centro de investigación agrícola de algodón y fibras en Bénin, donde Guazuncho 2 INTA y Chaco 520 INTA presentaron mayor asociación con Deltapine 90 de origen EE.UU. Esta misma tendencia fue observada por Gómez (2007) en estudios de la diversidad genética de 71 entradas de algodón pertenecientes al Banco de Germoplasma de INTA utilizando marcadores SSR. La variabilidad fenotípica examinada a través del Análisis de Componentes Principales (ACP) en las dos campañas agrícolas mostraron diferencias significativas. Las diferencias pueden ser explicada por las condiciones ambientales contrastantes entre campañas agrícolas, el menor porcentaje de precipitaciones (47.7% menor) en la campaña 2015/2016 provocó condiciones de estrés por falta de agua, induciendo un crecimiento menor y uniforme en los caracteres de AP, NN, NRF, LME, CAI, TPF, FV, Boch y Fij% (Tabla 5). Las condiciones ambientales en la segunda campaña (2016/2017) fueron más apropiadas para el crecimiento y la expresión de la variabilidad (Figura 2). En otros estudios (Spoljaric et al., 2018a; Spoljaric et al., 2018b) observaron comportamiento similar en la variabilidad cuando caracterizaron 20 genotipos de algodón de diferentes países frente a estrés hídrico en condiciones controladas (cámara de cultivo) y semi-controladas (invernáculo) determinando que las diferencias genéticas entre los genotipos para las variables asociadas al uso del agua fueron bajas debido a la presencia de interacciones (Tratamiento*Genotipo).
En relación al ACP para la correlación significativa (Elipses de confianza= Alfa=0,05) de la variable categórica origen geográfico indicó que no existen diferencias significativas entre las medias de los grupos mostrando semejanza entre orígenes geográficos (Figura 3). Sin embargo, los genotipos de origen geográfico de Argentina presentaron una mayor asociación con los genotipos estadounidenses versus a los genotipos de China. La principal diferencia fue dada en un mayor crecimiento vegetativo y reproductivo (AP, NN, NRF, LME, CAI y TPF) en los genotipos de origen geográfico de China. Estos mostraron ser más indeterminados y poseer un crecimiento compensatorio. Es decir, ante la pérdida de órganos reproductivos prolongan la producción de flores, manteniendo la tasa de floración tardía y número de sitios fructíferos (Sadras, 1995). Asimismo, los genotipos de Argentina y EE.UU. definieron su crecimiento más temprano. Esta característica está asociada al tipo de sistema de producción entre los orígenes geográficos. El sistema de producción en China en la mayoría de su área se basa en superficies pequeñas y cosecha manual, mientras que en Argentina y los EE.UU. el sistema de producción es a gran escala y cosecha mecanizada definiendo genotipos con adaptación local.
Los grupos se efectuaron por campaña agrícola y la ordenación de los mismos fue diferente en cada campaña, indicando que las condiciones ambientales y las respuestas de los caracteres fenotípicos presentaron mayor peso para la formación de los grupos y no su origen geográfico ya que en los diferentes grupos se encuentran entradas de orígenes geográficos distintos. Sin embargo, se pueden seleccionar los genotipos BGSP 756, BGSP 767 y BGSP 783 provenientes de China ya que en ambas campañas presentaron mayores distancias fenotípicas a los genotipos de Argentina y los EE.UU. Asimismo, presentaron mayor valor a la media general en las variables FV y Boch por planta en condiciones de estrés (Campaña 2015/2016), mientras que en la segunda campaña presentaron mayor valor promedio en características productivas de NRF y Fij%. Según Iqbal et al. (2005) y Wang et al. (2007) un mayor número de cápsulas por planta y el número de ramas fructíferas en la relación a las vegetativas, al igual que la precocidad en la emisión de ramas reproductivas, resultan de gran importancia para la obtención de cultivares más productivos. Además, estos resultados se condicen con el dendrograma obtenido a partir del análisis de SSR, donde se evidencia que los genotipos provenientes de China se diferencian del germoplasma argentino.
Si bien a nivel morfológico no se evidenciaron diferencias significativas entre los genotipos evaluados en las dos campañas, a nivel molecular los genotipos se separaron mostrando una tendencia a agruparse por su origen geográfico. El germoplasma de INTA evidenció menor distancia genética con el material de Deltapine 16 (EE.UU.) y mayor distancia genética con el de origen de China. En consecuencia, a partir de estos resultados se propone en investigaciones futuras realizar un diseño de combinaciones de cruzamientos entre los genotipos seleccionados y material elite del programa de mejora de INTA con el fin de evaluar la posible presencia de vigor hibrido en aquellos caracteres involucrados en la producción de este cultivo.
ABDALLA, A.; REDDY, O.; EL-ZIK, K. Y PEPPER, A. (2001). Genetic diversity and relationships of diploid and tetraploid cottons revealed using AFLP. Theor Appl Genet. 102:222–229.
BALZARINI, M. Y DI RIENZO, J. Info-Gen versión (2016). FCA, Universidad Nacional de Córdoba, Argentina. URL http://www.info-gen.com.ar
BALZARINI, M.; BRUNO, C.; PEÑA, A.; TEICH, I. Y DI RIENZO, J. (2010). Estadística en Biotecnología en Info-gen. Encuentro grupo Editor. Córdoba, Argentina.
BOTSTEIN, D.; WHITE, R.; SKOLNICK, M. Y DAVIS, R. (1980). Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American journal of human genetics, 32(3), 314–331.
BRUBAKER, C.; PATERSON, A. Y WENDEL, J. (1999). Comparative genetic mapping of allotetraploid cotton and its diploid progenitors. Genome 42: 184–203.
CHEN, G. Y DU, X. (2006). Genetic Diversity of Source Germplasm of Upland Cotton in China as Determined by SSR Marker Analysis Elsevier. Issue 8., 33 (8):733–745.
DJABOUTOU, M.; SINHA, M.; HOUEDJISSIN, S.; CACAI, G. Y AHANHANZO, C. (2017). Variability and Heritability of Morphological Traits in Collection of Cotton Genotypes (Gossypium hirsutum L.) and Their Potential Use for the Selection. European Scientific Journal, ESJ, 13(3), 385. https://doi.org/10.19044/esj.2017.v13n3p385
ENDRIZZI, J.; TURCOTTE, E. Y KOHEL, R. (1985). Genetics, cytology, and evolution of Gossypium. E.W. Caspari, G.S. John (Eds.), Advances in Genetics, Academic Press, 271-375p.
ETCHART, J. (2009). Detección de polimorfismos moleculares en materiales de Algodón (Gossypium hirsutum L.) fenotípicamente contrastante para la enfermedad azul y su mapeo en una población segregante. [Tésis Magister] Universidad Nacional de Rosario, Argentina, 122 p. https://fcagr.unr.edu.ar/?page_id=142
FANG, D.; HINZE, L.; PERCY, R.; LI, P.; DENG, D. Y THYSSEN, G. (2013). A microsatellite based genome wide analysis of genetic diversity and linkage disequilibrium in Upland cotton (Gossypium hirsutum L.) cultivars from major cotton-growing countries. Euphytica. 191:391–401.
GARRIS, A.; TAI, T.; COBURN, J.; KRESOVICH, S. Y MCCOUCH S. (2005). Genetic structure and diversity in Oryza sativa L. Genetics. 169:1631–1638. [PMC free article] [PubMed].
GENSUS GENETICA SUSTENTABLE: https://gensus.com.ar/productos/.
GIRBAL-BLACHA, N. (2012). La industria invisible: entre las finanzas y la política, empresas de cultura popular en la Argentina peronista: 1946-1955. H-industri@ (B. Aires) Vol. 06 Nro. 11
GÓMEZ, G. (2007). Estimación de la diversidad genética mediante marcadores Microsatélites en entradas de Algodón (Gossypium hirsutum L.) del Banco de germoplasma del INTA. [Tesis Magister] Universidad Nacional de Rosario, Argentina, 80 p.
IRCT, 1946. Institut de recherches du coton et des textiles exotiques. Paris, Francia.
IQBAL, Z.; LATEEF, M.; JABBAR, A.; MUHAMMAD, G. Y KHAN, M. (2005). Anthelmintic activity of Calotropis procera (Ait.) F. flowers in sheep. J. Ethnopharm., 102 (2): 256-261
JENKINS, H. (2003). Transmedia
storytelling: Moving characters from books to films to video games can make
them stronger and more compelling. MIT Technology.Review.
http://www.technologyreview.com/news/401760/transmedia-storytelling/
LAM, H.; XU, X.; LIU, X.; CHEN, W.; YANG, G.; WONG, F.; LI, M.; HE, W.; QIN, N. Y WANG, B. (2010). Resequencing of 31 wild and cultivated soybean genomes identifies patterns of genetic diversity and selection. Nat Genet. 42:1053–1059. [PubMed].
LI, Y.; LI, W.; ZHANG, C.; YANG, L.; CHANG, R.; GAUT, B. AND QIU LJ. 2010. Genetic diversity in domesticated soybean (Glycine max) and its wild progenitor (Glycine soja) for simple sequence repeat and single-nucleotide polymorphism loci. New Phytol. 188:242–253. [PubMed].
LIU, A. Y BURKE, J. (2006). Patterns of nucleotide diversity in wild and cultivated sunflower. Genetics. 173:321–330. [PMC free article] [PubMed]
LORI, L.; HINZE, E.; GAZAVE, M.; GORE, A.; FANG, D.; SCHEFFLER, B.; YU, J.; JONES, D.; FRELICHOWSKI, J. Y PERCY, R. (2016). Diversidad genética de las dos especies comerciales de algodón tetraploide en Gossypium Diversity Reference Set, Journal of Heredity, volumen 107, número 3, mayo de 2016, páginas 274–286, https://doi.org/10.1093/jhered/esw004
NILES, G. (1980). Plant breeding and improvement of the cotton plant. SAGE Journals 10 (4): 152-158
PEREIRA, G.; SOUSA, R.; HOFFMANN, L.; SILVA, E. Y BARROSO, P. (2012). Selective fertilization in interspecific crosses of allotetraploid species of Gossypium. Botany, v.90, p.159‑166, 2012. DOI: 10.1139/b11‑094
Programa de Asistencia para el Mejoramiento de la Calidad de la Fibra de Algodón (PROCALGODON) (2008). Ex Secretaria de Agricultura, Ganadería, Pesca y Alimentos.
R, DEVELOPMENT CORE TEAM. (2011). R: a language and environment for statistical computing. R Foundation for Statistical Computing. http://www.R-project.org/ verificado: abril de 2011
RAI R, et al. (2013). Gln3 mutations dissociate responses to nitrogen limitation (nitrogen catabolite repression) and rapamycin inhibition of TorC1. J Biol Chem 288(4):2789-804
ROYO, O.; POISSON, J.; BONACIC, I.; MONTENEGRO, A.; IBALO, S.; MAZZA, S. Y GIMÉNEZ, L. (2006). Plant Breeding and Genetics 20pg. https://inta.gob.ar/documentos/direction-of-cotton-breeding-in-argentina
SADRAS, V. (1995). Compensatory growth in cotton after loss of reproductive organs Field Crops Res., 40:.1-18.
SPOLJARIC, M.; TCACH, M.; ROJAS, J.; TARRAGO, J. Y COINTRY, E. (2018a). Caracterización de genotipos de Gossypium hirsutum L. sobre su tolerancia a estrés hídrico Recibido 09 de febrero de 2017 Publicado online 10 de abril de 2018 http://ria.inta.gob.ar/sites/default/files/trabajosenprensa/spoljaric-castellano-4.pdf
SPOLJARIC, M.; TCACH, M.; ROJAS, J.; TARRAGO, J. Y COINTRY, E. (2018b). Caracterización de genotipos de algodón por su tolerancia a estrés hídrico a través de variables asociadas al uso del agua. Revista RG News 4 (2) - Sociedade Brasileira de Recursos Genéticos. http://recursosgeneticos.org/Recursos/Arquivos/3._Caracterizaci_n_de_genotipos_de_algod_n_por_su_tolerancia_a_estr_s_h_drico_a_trav_s_de_variables_asociadas_al_uso_del_agua.pdf
TYAGI, P.; GORE, M.; BOWMAN, D.; CAMPBELL, B.; UDALL, J. Y KURAPARTHY, V. (2014). Genetic diversity and population structure in the US Upland cotton (Gossypium hirsutum L.). Theor Appl Genet. 127:283–295. [PubMed].
VIGOUROUX, Y.; MITCHELL, S.; MATSUOKA, Y.; HAMBLIN, M.; KRESOVICH, S.; SMITH, J.; JAQUETH, J.; SMITH, O. Y DOEBLEY, J. (2005). An analysis of genetic diversity across the maize genome using microsatellites. Genetics. 169:1617–1630. [PMC free article] [PubMed].
WANG, J.; AGRAWALA, M. Y COHEN, M. (2007) "Soft scissors: An interactive tool for realtime high quality matting", SIGGRAPH'07.
WENDEL, J.; BRUBAKER, C. Y PERCIVAL, A. (1992). Genetic diversity in Gossypium hirsutum and the origin of upland cotton. Am J Bot. 79:1291–1310.
WENDEL, J., BRUBAKER, C., Y SEELANAN, T. (2010). The origin and evolution of Gossypium. In JM. Stewart, D. Oosterhuis, J. J. Heithholt, & J. R. Mauney (Eds.), Physiology of cotton (pp. 1–18). Dordrecht, The Netherlands: Springer.
YU J., JUNG S., CHENG CH., LEE T., ZHENG P., BUBLE K., CRABB J., HUMANN J., HOUGH H., JONES D., CAMPBELL J., UDALL J., MAIN D. (2021). CottonGen: la base de datos comunitaria para la genómica del algodón, genética, y la investigación de mejoramiento. plantas 2021; 10(12):2805. https://www.cottongen.org/